Загрузите данные секвенирования в базу данных CNCB (Национальный центр биологической информации).
Данные секвенирования можно загрузить в CNCB (Китайский национальный центр биоинформации, Национальный центр биоинформации), который является собственной базой данных Китая и напрямую заменяет NCBI. По сравнению с NCBI, многие из его функций малоизвестны многим ученым. Поэтому здесь мы познакомим вас с использованием этих данных!
1. Официальный сайт
https://www.https://ngdc.cncb.ac.cn/gsub/https://ngdc.cncb.ac.cn/gsub/
2. Зарегистрированные пользователи
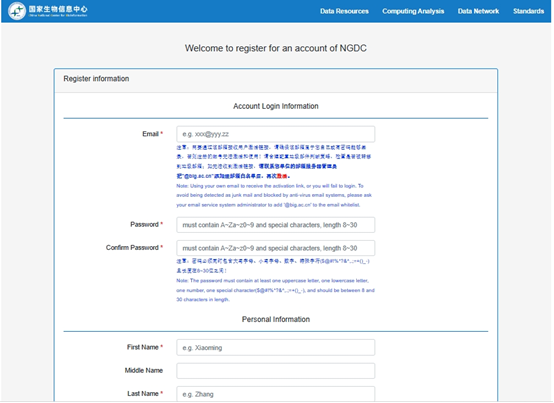
Нажмите Войти,Войдите в интерфейс регистрации,если ты не можешь найти,Затем перейдите непосредственно по этому URL-адресу.:https://ngdc.cncb.ac.cn/account/register?service=https://ngdc.cncb.ac.cn/gsub/login
Просто заполните его правдиво (обратите внимание: все должно быть заполнено на английском языке, адрес электронной почты должен быть способен получать электронные письма, а на следующем шаге потребуется проверка). После отправки регистрации просто подтвердите ее по электронной почте в течение 24 часов.
3. Войти
После завершения регистрации вернитесь на главную страницу для входа в свою учетную запись. После входа интерфейс выглядит следующим образом:

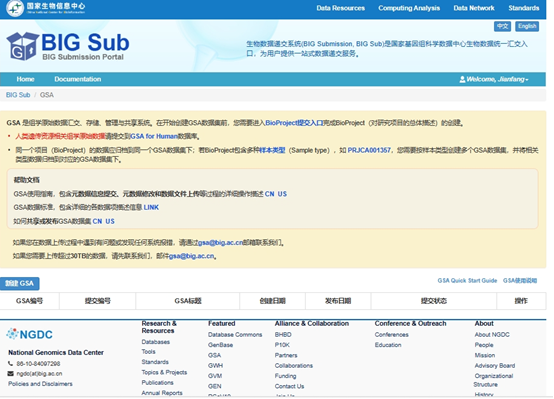
4. Загрузите данные
Нажмите «Архив последовательностей генома», чтобы войти в интерфейс загрузки данных.
4.1 Создать биопроект
Сначала нажмите «Портал подачи биопроекта», чтобы создать файл биопроекта;
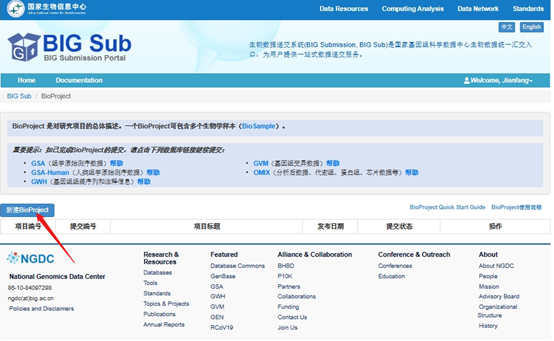
Нажмите, чтобы создать новый биопроект. Здесь вы можете изменить соответствующую информацию. По умолчанию система использует информацию, которую вы зарегистрировали при регистрации. После изменения нажмите «Сохранить» и перейдите к следующему шагу.
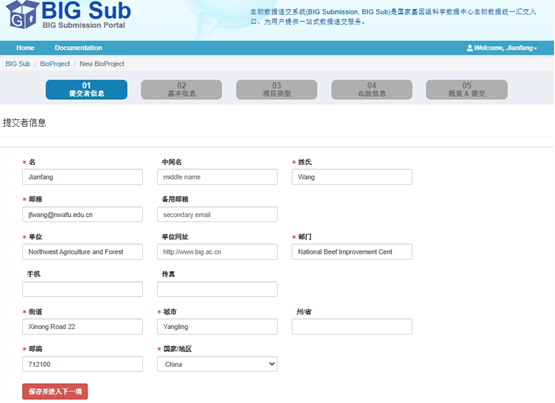
После выполнения пяти шагов нажмите «Отправить». Статус после отправки показан на рисунке ниже.

Более конкретные операции см. в руководстве: Вы можете просмотреть руководство по BioProject.

4.2 Создание биообразца (несколько биологических образцов)
Нажмите, чтобы создать новый биообразец (https://ngdc.cncb.ac.cn/gsub/submit/biosample/list), чтобы войти в интерфейс создания биообразца;
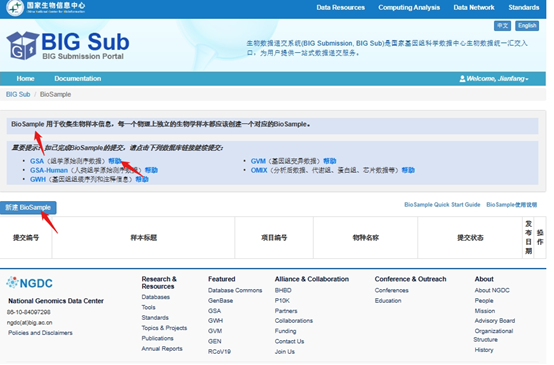
Заполняем информацию последовательно согласно подсказкам, но стоит отметить, что в режиме пакетной подачи проб (Batch BioSamples) (рекомендуется) нам необходимо загрузить новую сводную таблицу проб;
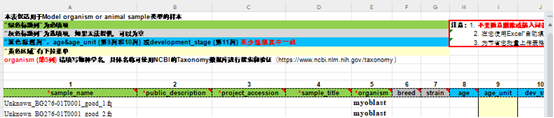
Если вы не знаете, как его заполнить, сначала скачайте кейс. Кейс очень понятен и проблем в принципе не будет;
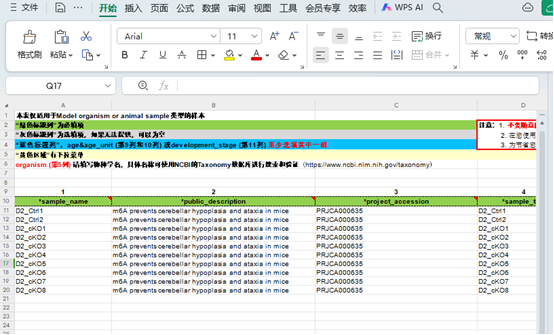
После заполнения нажмите «Отправить».
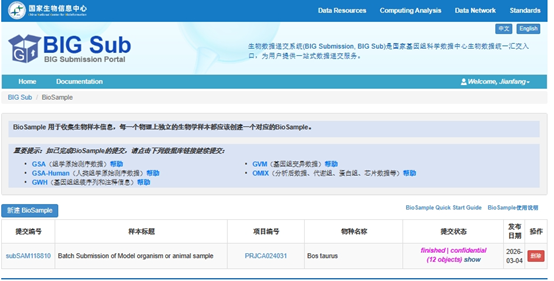
4.3 Отправка данных GSA (необработанная секвенация РНК)
После заполнения приложений BioProject и BioSample вы можете начать загрузку базы данных GSA. Подготовьте исходные данные, обычно с суффиксом .gz или .bz2. Сначала вернитесь в BIG Sub, снова выберите «Архив последовательностей генома» и войдите в исходный интерфейс загрузки данных. Трехэтапный метод работы резюмируется следующим образом:
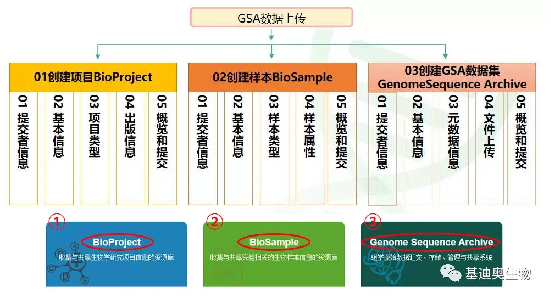
Создайте новый GSA:
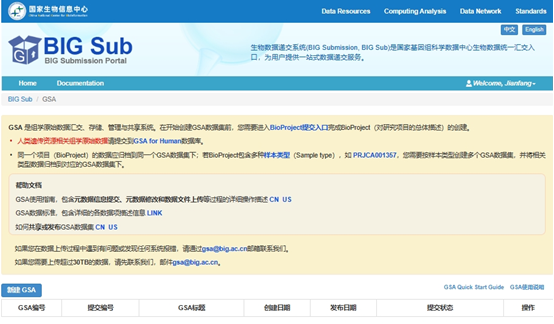
Следуйте подсказкам и заполняйте содержимое один за другим. На третьем этапе вам необходимо загрузить информацию о файле метаданных. Теперь мы загрузим шаблон заполнения и кейс.

Заполните информацию в Выражении:
Идентификатор в первом столбце должен начинаться с буквы E, например E1, E2, E3.... Для входа в BioProject необходимо ввести номер PRJCAxxxxx, который был передан на первом этапе подачи заявки на Biosample; имя должно быть таким же, как у того, кто подал заявку на биообразец. Имя_образца такое же; остальные заполняются в соответствии с платформой секвенирования или подсказкой.
Введите файл последовательности и информацию о коде MD5 в Run:
Формат данных секвенирования платформы Illumina обычно представляет собой файлы fastq (поддерживаются форматы сжатия gzip и bzip2). Проверка MD5. Как правило, после секвенирования компания предоставляет программное обеспечение MD5, и вы можете самостоятельно импортировать необработанные данные секвенирования и генерировать их автоматически.
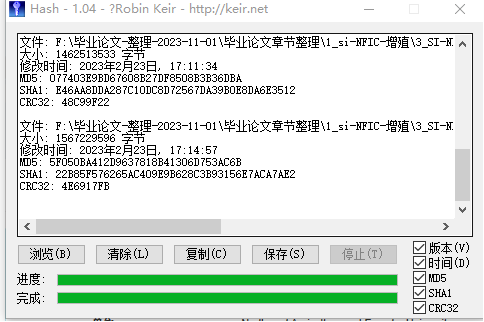
После того, как данные подготовлены, их можно загрузить. После загрузки нажмите «Проверить», чтобы проверить успешность загрузки данных:
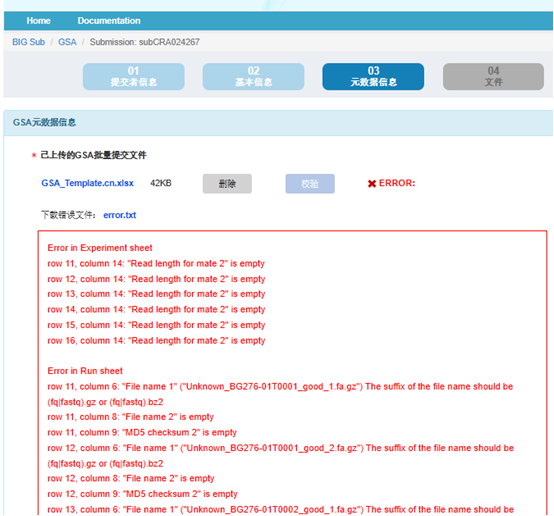
Если информация, заполненная в каком-либо столбце, неверна, система определит это и выдаст сообщение об ошибке. Мы можем следовать этому запросу, чтобы заполнить информацию в форме.
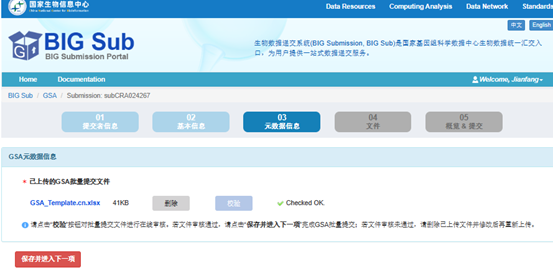
Далее выбираем метод загрузки. Здесь выбираем FTP для загрузки данных:
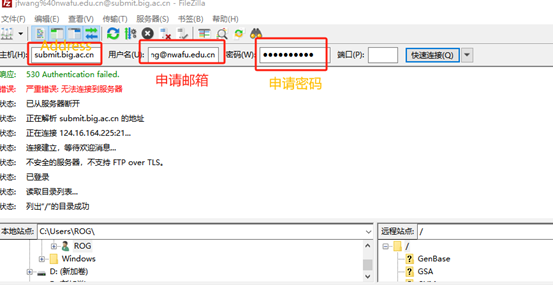
① Установите флажок FTP и запишите запрос на загрузку по FTP (рис. 3).

②Установите соединение. Откройте программное обеспечение и введите информацию о хосте как «submit.big.ac.cn». Имя пользователя и пароль соответствуют адресу электронной почты и паролю учетной записи для входа в базу данных GSA. Нажмите «Быстрое подключение», и в строке состояния отобразится успешный вход.
③Введите каталог загрузки. После успешного входа в систему выберите локальный путь, соответствующий данным, которые будут загружены в «Локальный сайт». В «Удаленном сайте» щелкните папку GSA, чтобы войти в каталог GSA (не загружайте файл в корневой каталог, иначе программа фоновой обработки не сможет сканировать загружаемый файл).
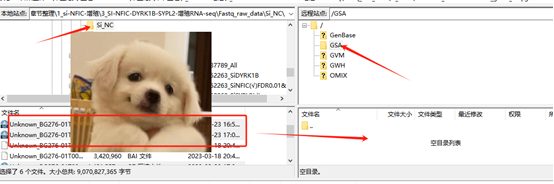
Проверьте скорость загрузки, и в каталоге удаленного сайта появятся соответствующие данные, указывающие на успешную передачу данных.

Загрузите данные в каталог GSA. Рекомендуется создать подкаталог для каждого пакета данных для хранения данных.

После успешной загрузки нажмите, чтобы перейти к следующему шагу.

После завершения загрузки вы можете еще раз проверить информацию и отправить ее после подтверждения ее правильности. Пожалуйста, терпеливо ждите проверки! Разве это не очень просто? Каждый может оставить сообщение!

Неразрушающее увеличение изображений одним щелчком мыши, чтобы сделать их более четкими артефактами искусственного интеллекта, включая руководства по установке и использованию.

Копикодер: этот инструмент отлично работает с Cursor, Bolt и V0! Предоставьте более качественные подсказки для разработки интерфейса (создание навигационного веб-сайта с использованием искусственного интеллекта).

Новый бесплатный RooCline превосходит Cline v3.1? ! Быстрее, умнее и лучше вилка Cline! (Независимое программирование AI, порог 0)

Разработав более 10 проектов с помощью Cursor, я собрал 10 примеров и 60 подсказок.

Я потратил 72 часа на изучение курсорных агентов, и вот неоспоримые факты, которыми я должен поделиться!
Идеальная интеграция Cursor и DeepSeek API

DeepSeek V3 снижает затраты на обучение больших моделей

Артефакт, увеличивающий количество очков: на основе улучшения характеристик препятствия малым целям Yolov8 (SEAM, MultiSEAM).

DeepSeek V3 раскручивался уже три дня. Сегодня я попробовал самопровозглашенную модель «ChatGPT».

Open Devin — инженер-программист искусственного интеллекта с открытым исходным кодом, который меньше программирует и больше создает.

Эксклюзивное оригинальное улучшение YOLOv8: собственная разработка SPPF | SPPF сочетается с воспринимаемой большой сверткой ядра UniRepLK, а свертка с большим ядром + без расширения улучшает восприимчивое поле

Популярное и подробное объяснение DeepSeek-V3: от его появления до преимуществ и сравнения с GPT-4o.

9 основных словесных инструкций по доработке академических работ с помощью ChatGPT, эффективных и практичных, которые стоит собрать

Вызовите deepseek в vscode для реализации программирования с помощью искусственного интеллекта.

Познакомьтесь с принципами сверточных нейронных сетей (CNN) в одной статье (суперподробно)

50,3 тыс. звезд! Immich: автономное решение для резервного копирования фотографий и видео, которое экономит деньги и избавляет от беспокойства.

Cloud Native|Практика: установка Dashbaord для K8s, графика неплохая

Краткий обзор статьи — использование синтетических данных при обучении больших моделей и оптимизации производительности

MiniPerplx: новая поисковая система искусственного интеллекта с открытым исходным кодом, спонсируемая xAI и Vercel.

Конструкция сервиса Synology Drive сочетает проникновение в интрасеть и синхронизацию папок заметок Obsidian в облаке.

Центр конфигурации————Накос

Начинаем с нуля при разработке в облаке Copilot: начать разработку с минимальным использованием кода стало проще

[Серия Docker] Docker создает мультиплатформенные образы: практика архитектуры Arm64

Обновление новых возможностей coze | Я использовал coze для создания апплета помощника по исправлению домашних заданий по математике

Советы по развертыванию Nginx: практическое создание статических веб-сайтов на облачных серверах

Feiniu fnos использует Docker для развертывания личного блокнота Notepad

Сверточная нейронная сеть VGG реализует классификацию изображений Cifar10 — практический опыт Pytorch

Начало работы с EdgeonePages — новым недорогим решением для хостинга веб-сайтов

[Зона легкого облачного игрового сервера] Управление игровыми архивами


