Точное воспроизведение литературы — объект seurat добавляет метаинформацию о клеточных субпопуляциях.
❝Я продолжал изучать набор данных научных твитов, который появился в твитах на прошлой неделе, и продолжу писать о твитах на этой неделе. ❞
Название статьи:
CXCL9:SPP1 macrophage polarity identifies a network of cellular programs that control human cancers.
Science:

Набор данных: GSE234933
Во всем твите следует отметить три момента:
- Данные, предоставленные автором, представляют собой комбинацию нескольких файлов формата rds, сжатых вместе.,После распаковки прочитайте файлы в цикле и объедините их в seuratобъект.
- Информация о субпопуляции ячеек, предоставленная автором, может быть впоследствии добавлена к информации метаданных, чтобы ее можно было напрямую назвать в честь ее ячеек.
- Проверьте статус группировкикогда,Поскольку была добавлена информация о субпопуляции клеток.,Однако, поскольку автор отфильтровал некоторые ячейки на ранней стадии,,В итоге осталось всего 187 399 ячеек.,Поэтому часть NA необходимо удалить.
# install.packages('R.utils')
rm(list=ls())
options(stringsAsFactors = F)
library(Seurat)
library(ggplot2)
library(clustree)
library(cowplot)
library(dplyr)
library(data.table)
library(stringr)
#install.packages("textshape")
library(textshape)
#setwd("../../")
# Получить список всех файлов rds
file_list <- list.files("./GSE234933_raw/rds/", pattern = ".rds")
# Создайте пустой список для хранилищаSeuratобъектов.
seurat_list <- list()
# Цикл для чтения данных каждого файла rds и создания Seuratобъекта.
for (file in file_list) {
# Путь к файлу сращивания
data.path <- paste0("./GSE234933_raw/rds/", file)
# Чтение данных файлов RDS
seurat_data <- readRDS(data.path)
# Создайте Seuratобъект и укажите имя проекта в качестве имени файла (удалите суффикс)
sample_name <- tools::file_path_sans_ext(basename(file))
seurat_obj <- CreateSeuratObject(counts = seurat_data,
project = sample_name,
min.features = 200,
min.cells = 3)
# Добавить Seuratобъект в список
seurat_list <- append(seurat_list, seurat_obj)
}
# Извлеките часть перед подчеркиванием
sample_names <- sub("_.*", "", file_list)
# Объединить объекты Seurat, объединить все объекты Seurat в один объект
seurat_combined <- merge(seurat_list[[1]],
y = seurat_list[-1])
# Распечатать объединенный объект Seurat
print(seurat_combined)
seurat_combined@assays$RNA@counts[1:10, 1:2]
##хранилищеданные Это займет больше времени####
saveRDS(seurat_combined, file = "seurat2.rds")
Прочитайте информацию о субпопуляции ячеек, заданную набором данных.
###Прочитайте информацию о субпопуляции ячеек, заданную набором данных.
cell<-fread("./GSE234933_raw/GSE234933_MGH_HNSCC_cell_annotation.txt.gz")
colnames(cell)
meta2 <- cell %>% column_to_rownames("sample_barcode")
###Два метода могут добавлять информацию о подгруппе ячеек, столбец подкластеризации, указанный в статье.
# sce_obj <- CreateSeuratObject(counts = seurat_combined@assays$RNA$counts,
# meta.data =meta2,
# project = sample_name,
# min.features = 200,
# min.cells = 3)
sce3 <- AddMetaData(object = seurat_combined, metadata = meta2)
head(sce3@meta.data)
Группировка и гармония уменьшения размерности
###########
###Группировка и гармония уменьшения размерности
dir.create("2-harmony2")
getwd()
setwd("2-harmony2")
# sce.all=readRDS("../1-QC/sce.all_qc.rds")
sce=sce3
sce
sce <- NormalizeData(sce,
normalization.method = "LogNormalize",
scale.factor = 1e4)
sce <- FindVariableFeatures(sce)
sce <- ScaleData(sce)
sce <- RunPCA(sce, features = VariableFeatures(object = sce))
#install.packages("harmony")
library(harmony)
seuratObj <- RunHarmony(sce, "orig.ident")
names(seuratObj@reductions)
seuratObj <- RunUMAP(seuratObj, dims = 1:15,
reduction = "harmony")
DimPlot(seuratObj,reduction = "umap",label=T )
sce=seuratObj
sce <- FindNeighbors(sce, reduction = "harmony",
dims = 1:15)
sce.all=sce
sce.all=FindClusters(sce.all, #graph.name = "CCA_snn",
resolution = 0.05, algorithm = 1)
sel.clust = "RNA_snn_res.0.05"
sce.all <- SetIdent(sce.all, value = sel.clust)
table(sce.all@active.ident)
saveRDS(sce.all, "sce.all_int0.05.rds")
Проверьте статус группировки
####################
#####Проверьте статус группировки
DimPlot(sce.all, reduction = "umap", group.by = "seurat_clusters",label = T)
colnames(sce.all@meta.data)
DimPlot(sce.all, reduction = "umap", group.by = "subclustering",label = T)
##### Подгруппа с удаленной частью NA#######
table(sce.all$subclustering)
###подгруппировать<NA>Назначение0
sce.all$subclustering = sce.all$subclustering %>%
replace(x = ., list =is.na(.), values =0)
sce.all$celltype<-ifelse(sce.all$subclustering=="Endothelial cells","Endothelial cells",
ifelse(sce.all$subclustering=="Fibroblasts","Fibroblasts",
ifelse(sce.all$subclustering=="Lymphocytes","Lymphocytes",
ifelse(sce.all$subclustering=="Myeloid cells","Myeloid cells",
ifelse(sce.all$subclustering=="Tumor cells","Tumor cells","NA")))))
table(sce.all$celltype)
sce <- sce.all[, !(sce.all$celltype %in% c('NA'))]
DimPlot(sce, reduction = "umap", group.by = "celltype",label=T)
ggsave('umap_by_RNA_snn_res.0.05_paper.pdf',width =7,height = 6)
«Картинка слева — это картинка в статье, а картинка справа — то, что я получил после добавления метаинформации с последующим уменьшением размерности и повторной группировкой. Возможность воспроизведения очень высока».
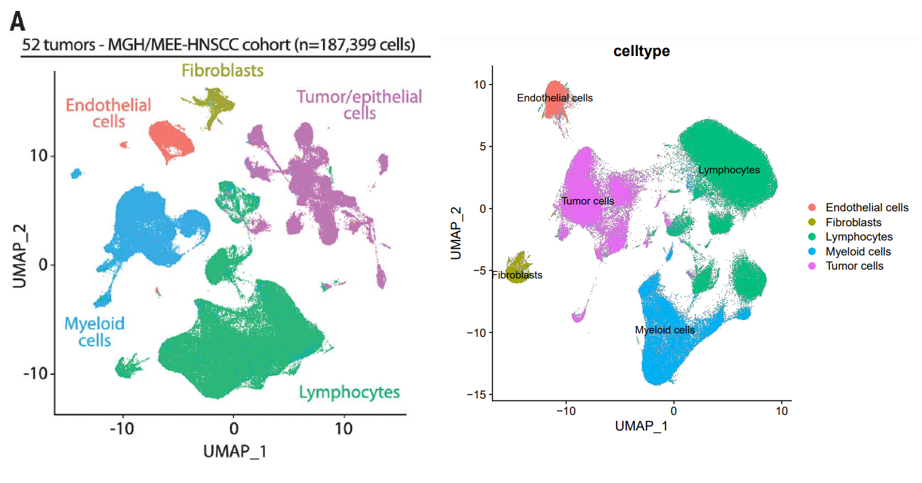
«Результаты, полученные таким способом, будут более удобными, если мы продолжим воспроизводить последующий анализ. Хотя в последнем твите также были разделены подгруппы клеток, у разных людей все же разное понимание подразделения подгрупп.

Неразрушающее увеличение изображений одним щелчком мыши, чтобы сделать их более четкими артефактами искусственного интеллекта, включая руководства по установке и использованию.

Копикодер: этот инструмент отлично работает с Cursor, Bolt и V0! Предоставьте более качественные подсказки для разработки интерфейса (создание навигационного веб-сайта с использованием искусственного интеллекта).

Новый бесплатный RooCline превосходит Cline v3.1? ! Быстрее, умнее и лучше вилка Cline! (Независимое программирование AI, порог 0)

Разработав более 10 проектов с помощью Cursor, я собрал 10 примеров и 60 подсказок.

Я потратил 72 часа на изучение курсорных агентов, и вот неоспоримые факты, которыми я должен поделиться!
Идеальная интеграция Cursor и DeepSeek API

DeepSeek V3 снижает затраты на обучение больших моделей

Артефакт, увеличивающий количество очков: на основе улучшения характеристик препятствия малым целям Yolov8 (SEAM, MultiSEAM).

DeepSeek V3 раскручивался уже три дня. Сегодня я попробовал самопровозглашенную модель «ChatGPT».

Open Devin — инженер-программист искусственного интеллекта с открытым исходным кодом, который меньше программирует и больше создает.

Эксклюзивное оригинальное улучшение YOLOv8: собственная разработка SPPF | SPPF сочетается с воспринимаемой большой сверткой ядра UniRepLK, а свертка с большим ядром + без расширения улучшает восприимчивое поле

Популярное и подробное объяснение DeepSeek-V3: от его появления до преимуществ и сравнения с GPT-4o.

9 основных словесных инструкций по доработке академических работ с помощью ChatGPT, эффективных и практичных, которые стоит собрать

Вызовите deepseek в vscode для реализации программирования с помощью искусственного интеллекта.

Познакомьтесь с принципами сверточных нейронных сетей (CNN) в одной статье (суперподробно)

50,3 тыс. звезд! Immich: автономное решение для резервного копирования фотографий и видео, которое экономит деньги и избавляет от беспокойства.

Cloud Native|Практика: установка Dashbaord для K8s, графика неплохая

Краткий обзор статьи — использование синтетических данных при обучении больших моделей и оптимизации производительности

MiniPerplx: новая поисковая система искусственного интеллекта с открытым исходным кодом, спонсируемая xAI и Vercel.

Конструкция сервиса Synology Drive сочетает проникновение в интрасеть и синхронизацию папок заметок Obsidian в облаке.

Центр конфигурации————Накос

Начинаем с нуля при разработке в облаке Copilot: начать разработку с минимальным использованием кода стало проще

[Серия Docker] Docker создает мультиплатформенные образы: практика архитектуры Arm64

Обновление новых возможностей coze | Я использовал coze для создания апплета помощника по исправлению домашних заданий по математике

Советы по развертыванию Nginx: практическое создание статических веб-сайтов на облачных серверах

Feiniu fnos использует Docker для развертывания личного блокнота Notepad

Сверточная нейронная сеть VGG реализует классификацию изображений Cifar10 — практический опыт Pytorch

Начало работы с EdgeonePages — новым недорогим решением для хостинга веб-сайтов

[Зона легкого облачного игрового сервера] Управление игровыми архивами


