RNAseq|Mime-версия кода — идеальное сочетание 101 алгоритма машинного обучения для построения оптимальной прогностической модели.
Mime1 предоставляет удобное решение для построения ансамблевых моделей на основе машинного обучения с использованием сложных наборов данных для идентификации ключевых генов, связанных с прогнозом.
Ранее вводился отдельноLasso ,randomForestSRC,Enet(Elastic Net),CoxBoost и SuperPC При построении параметров метода Моделиз выживания в этой статье рассказывается, как использовать один стиль пакета Mime1 для завершения машинного описания в литературе. обучениекомбинацияиз анализа и вывода уровня документа из диаграммы.
Кроме того, дополнительное введениеодин Вниз(1) На что следует обратить внимание при замене собственных данных (2)какИзвлечение результатов оценки риска в соответствии с указанной модельюПровести последующий анализ и (3)какСравните модели для целевых типов рака(Глиому можно использоватьданныевстроенныйиз)(Лично я считаю, что это важнее)
1. Загрузите пакет R, данные
Этот пакет объединяет 10 типов пакетов машинного обучения, поэтому установка будет немного сложной. Наберитесь терпения и загрузите все, что вам нужно.
# options("repos"= c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
# options(BioC_mirror="http://mirrors.tuna.tsinghua.edu.cn/bioconductor/")
if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager")
depens<-c('GSEABase', 'GSVA', 'cancerclass', 'mixOmics', 'sparrow', 'sva' , 'ComplexHeatmap' )
for(i in 1:length(depens)){
depen<-depens[i]
if (!requireNamespace(depen, quietly = TRUE)) BiocManager::install(depen,update = FALSE)
}
if (!requireNamespace("CoxBoost", quietly = TRUE))
devtools::install_github("binderh/CoxBoost")
if (!requireNamespace("fastAdaboost", quietly = TRUE))
devtools::install_github("souravc83/fastAdaboost")
if (!requireNamespace("Mime", quietly = TRUE))
devtools::install_github("l-magnificence/Mime")
library(Mime1)Сначала посмотрите на пример данных
1. Данные генного набора
может бытьдифференциальные гены,Путь к точке доступа (MsigDB),WGCNAилиPPIоказатьсяизhub gene ,илиМаркерный ген определенного подтипа одиночных клетоки т. д. Целевой набор генов, полученный любыми способами
load("./External data/genelist.Rdata")
#> [1] "MYC" "CTNNB1" "JAG2" "NOTCH1" "DLL1" "AXIN2" "PSEN2" "FZD1" "NOTCH4" "LEF1" "AXIN1" "NKD1" "WNT5B"
#>[14] "CUL1" "JAG1" "MAML1" "KAT2A" "GNAI1" "WNT6" "PTCH1" "NCOR2" "DKK4" "HDAC2" "DKK1" "TCF7" "WNT1"
#>[27] "NUMB" "ADAM17" "DVL2" "PPARD" "NCSTN" "HDAC5" "CCND2" "FRAT1" "CSNK1E" "RBPJ" "FZD8" "TP53" "SKP2"
#>[40] "HEY2" "HEY1" "HDAC11"2. Данные о выживаемости и информация об экспрессии генов.
load("./External data/Example.cohort.Rdata") # Данные о выживании и информация об экспрессии генов
list_train_vali_Data[["Dataset1"]][1:5,1:5]
# ID OS.time OS MT-CO1 MT-CO3
#60 TCGA.DH.A66B.01 1281.65322 0 13.77340 13.67931
#234 TCGA.HT.7607.01 96.19915 1 14.96535 14.31857
#42 TCGA.DB.A64Q.01 182.37755 0 13.90659 13.65321
#126 TCGA.DU.8167.01 471.97707 0 14.90695 14.59776
#237 TCGA.HT.7610.01 1709.53901 0 15.22784 14.62756Среди них list_train_vali_Data — это список, содержащий два набора данных. Первый столбец каждого набора данных — это идентификатор, столбцы 2–3 — это информация о выживании (OS.time, OS), а следующий — экспрессия генов.
2. Построение прогностической модели.
1. Создайте комбинацию моделей машинного обучения из 101.
Этот пакет значительно снижает стоимость обучения и может быть создан непосредственно с помощью функции ML.Dev.Prog.Sig.
res <- ML.Dev.Prog.Sig(train_data = list_train_vali_Data$Dataset1,
list_train_vali_Data = list_train_vali_Data,
unicox.filter.for.candi = T,
unicox_p_cutoff = 0.05,
candidate_genes = genelist,
mode = 'all',nodesize =5,seed = 5201314 )ML.Dev.Prog.Sig()Необязательныйall,singleиdoubleтри видамодель.allдля всех 10 алгоритмов а также комбинация .singleдля использует изодин среди 10 алгоритмов.doubleдля двух алгоритмов изкомбинации обычно используется одинallмодель.- По умолчанию
unicox.filter.for.candiдляT, Однофакторный анализ Кокса сначала будет выполнен на обучающем наборе, unicox_p_cutoff. Значительные гены будут использованы в Постройке. прогностическую модель.
еслиПри использовании собственных данных,Необходимо обратить внимание на:
(1)заменить себяданные Уведомлениепервые три столбцаиз Требовать,и несколькоданные Собрать сформа спискахранилище。
(2) Лучше всего подтвердить это перед анализом. Во всех ли наборах данных В списке набора генов есть все гены. ,уменьшатьСообщить об ошибке。
(3) Если количество семян будет определено, это окажет небольшое влияние.
Проверив из любопытства, я обнаружил, что в образце данных Dataset2 отсутствуют несколько генов в наборе генов, но почему не сообщается об ошибке?
data2 <- data2 %>%
dplyr::select(ID , OS.time , OS, genelist )
#Error in `dplyr::select()`:
#! Can't select columns that don't exist.
#✖ Columns `JAG1`, `DKK4`, and `WNT1` don't exist.
#Run `rlang::last_trace()` to see where the error occurred.Проверьте функцию через View(ML.Dev.Prog.Sig) и установите unicox.filter.for.candi = T. Сначала будет выполнен однофакторный анализ Cox. В качестве генов-кандидатов для машины будут использоваться только гены со значимыми отдельными факторами. обучение 101 комбинации моделей. После последующего анализа гены, значимые для одного гена в красном поле на рисунке ниже, не содержат генов «JAG1», «DKK4» и WNT1, которые отсутствуют в наборе данных 2, поэтому ошибка не возникает. сообщил.
Однако если вы не уверены, какие гены важны для однофакторного прогноза, вам следует сначала убедиться, что обучающий набор и набор проверки содержат список генов и все гены при анализе собственных результатов.
2. Отображение C-индекса
Пример данных list_train_vali_Data представляет собой список из двух наборов данных. На изображении результатов есть две очереди. Последние два столбца — это средние значения Cindex. Это основное изображение в литературе по комбинациям моделей машинного обучения.
cindex_dis_all(res,
validate_set = names(list_train_vali_Data)[-1],
order = names(list_train_vali_Data),
width = 0.35
)3. Просмотрите результаты указанной модели.
Предположим, мы выбрали первую модель (StepCox[forward] + plsRcox), мы можем просмотреть производительность cindex каждого набора данных в рамках этой модели отдельно.
cindex_dis_select(res,
model="StepCox[forward] + plsRcox",
order= names(list_train_vali_Data))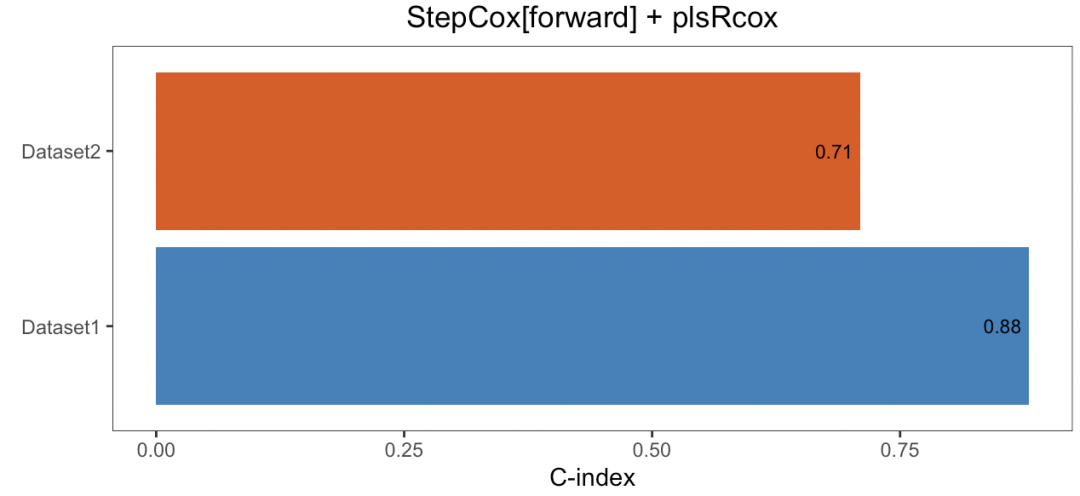
Вы также можете просмотреть кривые КМ каждого набора данных в рамках этой модели.
survplot <- vector("list",2)
for (i in c(1:2)) {
print(survplot[[i]]<-rs_sur(res, model_name = "StepCox[forward] + plsRcox",
dataset = names(list_train_vali_Data)[i],
#color=c("blue","green"),
median.line = "hv",
cutoff = 0.5,
conf.int = T,
xlab="Day",pval.coord=c(1000,0.9))
)
}
aplot::plot_list(gglist=survplot,ncol=2)Извлечение результатов модели RS
Очень важным моментом здесь является извлечение результатов РС по указанной модели, а затем вы можете перерисовать КМ и другие анализы, такие как независимый прогностический анализ, лесной график, номограмму и т. д. в соответствии с вашими потребностями.
Все результаты представлены в формате res. Вы можете узнать соответствующую информацию по str(res) и извлечь ее.
head(res$riskscore$`StepCox[forward] + plsRcox`[[1]])
head(res$riskscore$`StepCox[forward] + plsRcox`[[2]])4. Результаты AUC
Рассчитайте значения AUC за 1, 3 и 5 лет для каждой модели и визуализируйте результаты AUC за 1 год для всех моделей.
all.auc.1y <- cal_AUC_ml_res(res.by.ML.Dev.Prog.Sig = res,train_data = list_train_vali_Data[["Dataset1"]],
inputmatrix.list = list_train_vali_Data,mode = 'all',AUC_time = 1,
auc_cal_method="KM")
all.auc.3y <- cal_AUC_ml_res(res.by.ML.Dev.Prog.Sig = res,train_data = list_train_vali_Data[["Dataset1"]],
inputmatrix.list = list_train_vali_Data,mode = 'all',AUC_time = 3,
auc_cal_method="KM")
all.auc.5y <- cal_AUC_ml_res(res.by.ML.Dev.Prog.Sig = res,train_data = list_train_vali_Data[["Dataset1"]],
inputmatrix.list = list_train_vali_Data,mode = 'all',AUC_time = 5,
auc_cal_method="KM")
auc_dis_all(all.auc.1y,
dataset = names(list_train_vali_Data),
validate_set=names(list_train_vali_Data)[-1],
order= names(list_train_vali_Data),
width = 0.35,
year=1)Вы также можете нарисовать кривую AUC под выбранной моделью.
roc_vis(all.auc.1y,
model_name = "StepCox[forward] + plsRcox",
dataset = names(list_train_vali_Data),
order= names(list_train_vali_Data),
anno_position=c(0.65,0.55),
year=1)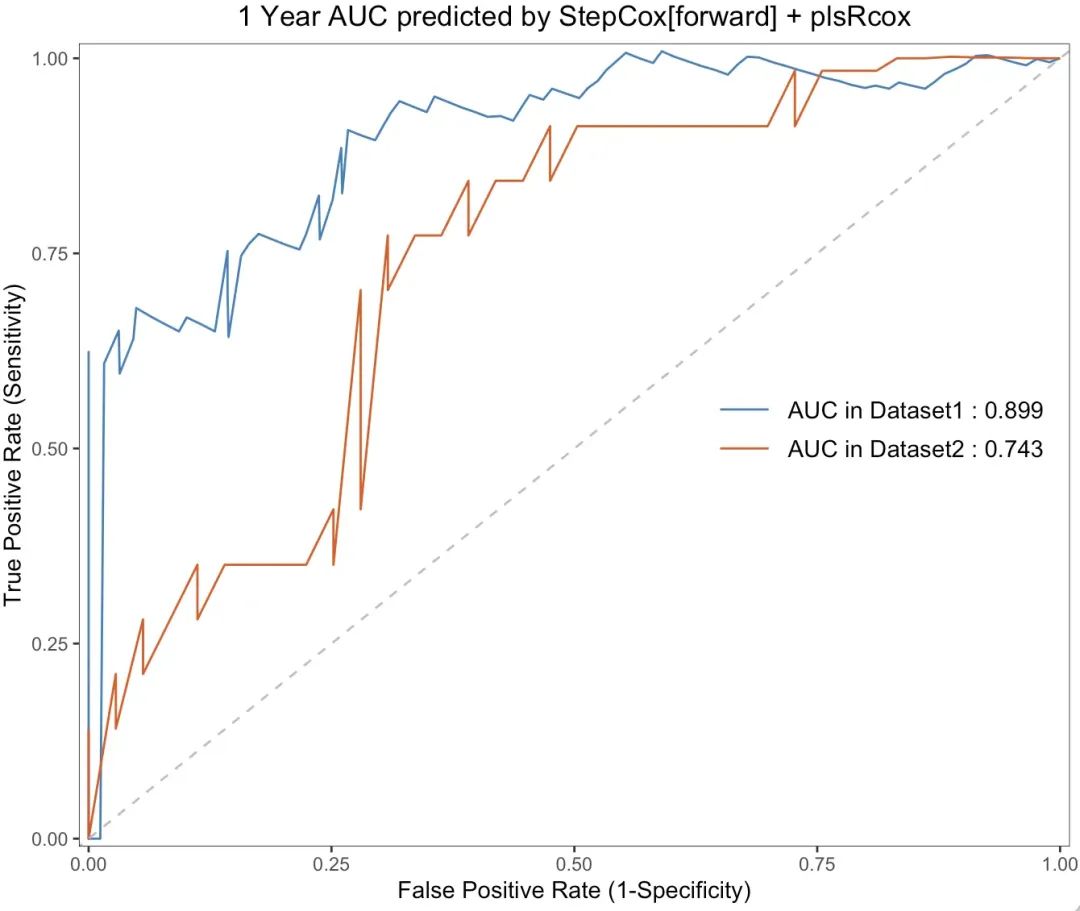
auc_dis_select(list(all.auc.1y,all.auc.3y,all.auc.5y),
model_name="StepCox[forward] + plsRcox",
dataset = names(list_train_vali_Data),
order= names(list_train_vali_Data),
year=c(1,3,5))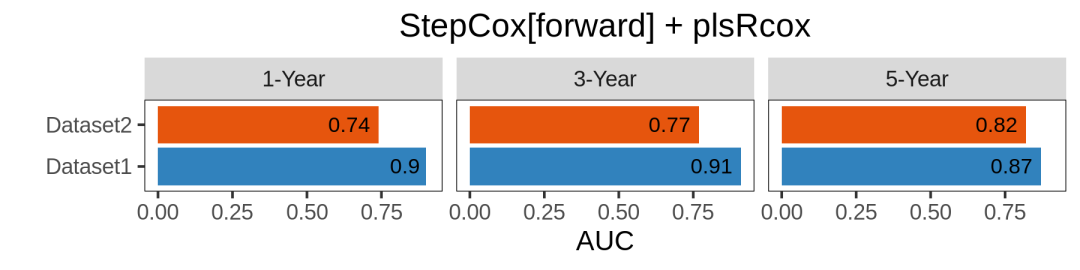
5. Сравнение моделей
Пакет также предоставляет прогностические функции, о которых ранее сообщалось в литературе, но, конечно, касается только глиом.
Что делать, если у вас другие виды рака?Вы можете увидеть, как это работает, посмотрев на функциюиз Форма ввода,Затем просто сделайте соответствующую замену и можно будет ее проанализировать. (очень важно)
cc.glioma.lgg.gbm <- cal_cindex_pre.prog.sig(use_your_own_collected_sig = F,
type.sig = c('Glioma','LGG','GBM'),
list_input_data = list_train_vali_Data)
cindex_comp(cc.glioma.lgg.gbm,
res,
model_name="StepCox[forward] + plsRcox",
dataset=names(list_train_vali_Data))Посмотрите на функцию, чтобы найти форму встроенной модели.
type.sig = c('Glioma','LGG','GBM')
pre.prog.sig <- Mime1::pre.prog.sig
if (all(type.sig %in% names(pre.prog.sig))) {
if (length(type.sig) == 1) {
sig.input <- pre.prog.sig[[type.sig[1]]]
}
else {
sig.input <- pre.prog.sig[[type.sig[1]]]
for (i in 2:length(type.sig)) {
sig.input <- rbind(sig.input, pre.prog.sig[[type.sig[i]]])
}
}
}
View(sig.input)Проверив документ 30810537, мы обнаружили, что гены (тепловая карта), которые в конечном итоге составляют оценку риска, согласуются с генами, встроенными в пакет, и коэффициенты на правом рисунке также согласуются.
Хорошо, теперь мы завершили построение и проверку прогностической модели. Позже нам может понадобиться извлечь модель RS в соответствии с содержанием статьи для независимого прогностического тестирования и некоторого визуального анализа.
Теперь все, что вам нужно сделать, это (1) Подготовить целевые виды рака из TCGAданные и GEOданные — используемые для построения прогноза 101Модель , (2) Список генов целевой прогностической модели рака а также Соответствующий коэффициент -- для сравнения модели. (3) Напишите статьи и опубликуйте их.。
Ссылки:https://github.com/l-magnificence/Mime?tab=readme-ov-file

Неразрушающее увеличение изображений одним щелчком мыши, чтобы сделать их более четкими артефактами искусственного интеллекта, включая руководства по установке и использованию.

Копикодер: этот инструмент отлично работает с Cursor, Bolt и V0! Предоставьте более качественные подсказки для разработки интерфейса (создание навигационного веб-сайта с использованием искусственного интеллекта).

Новый бесплатный RooCline превосходит Cline v3.1? ! Быстрее, умнее и лучше вилка Cline! (Независимое программирование AI, порог 0)

Разработав более 10 проектов с помощью Cursor, я собрал 10 примеров и 60 подсказок.

Я потратил 72 часа на изучение курсорных агентов, и вот неоспоримые факты, которыми я должен поделиться!
Идеальная интеграция Cursor и DeepSeek API

DeepSeek V3 снижает затраты на обучение больших моделей

Артефакт, увеличивающий количество очков: на основе улучшения характеристик препятствия малым целям Yolov8 (SEAM, MultiSEAM).

DeepSeek V3 раскручивался уже три дня. Сегодня я попробовал самопровозглашенную модель «ChatGPT».

Open Devin — инженер-программист искусственного интеллекта с открытым исходным кодом, который меньше программирует и больше создает.

Эксклюзивное оригинальное улучшение YOLOv8: собственная разработка SPPF | SPPF сочетается с воспринимаемой большой сверткой ядра UniRepLK, а свертка с большим ядром + без расширения улучшает восприимчивое поле

Популярное и подробное объяснение DeepSeek-V3: от его появления до преимуществ и сравнения с GPT-4o.

9 основных словесных инструкций по доработке академических работ с помощью ChatGPT, эффективных и практичных, которые стоит собрать

Вызовите deepseek в vscode для реализации программирования с помощью искусственного интеллекта.

Познакомьтесь с принципами сверточных нейронных сетей (CNN) в одной статье (суперподробно)

50,3 тыс. звезд! Immich: автономное решение для резервного копирования фотографий и видео, которое экономит деньги и избавляет от беспокойства.

Cloud Native|Практика: установка Dashbaord для K8s, графика неплохая

Краткий обзор статьи — использование синтетических данных при обучении больших моделей и оптимизации производительности

MiniPerplx: новая поисковая система искусственного интеллекта с открытым исходным кодом, спонсируемая xAI и Vercel.

Конструкция сервиса Synology Drive сочетает проникновение в интрасеть и синхронизацию папок заметок Obsidian в облаке.

Центр конфигурации————Накос

Начинаем с нуля при разработке в облаке Copilot: начать разработку с минимальным использованием кода стало проще

[Серия Docker] Docker создает мультиплатформенные образы: практика архитектуры Arm64

Обновление новых возможностей coze | Я использовал coze для создания апплета помощника по исправлению домашних заданий по математике

Советы по развертыванию Nginx: практическое создание статических веб-сайтов на облачных серверах

Feiniu fnos использует Docker для развертывания личного блокнота Notepad

Сверточная нейронная сеть VGG реализует классификацию изображений Cifar10 — практический опыт Pytorch

Начало работы с EdgeonePages — новым недорогим решением для хостинга веб-сайтов

[Зона легкого облачного игрового сервера] Управление игровыми архивами


