Обучение процессу анализа микро РНК (3)/предсказание целевого гена микро РНК - загрузка данных из базы данных ENCORI
После получения микро РНК вам нужно попытаться предсказать их цели.,Обычно мы будем использовать интегрированный анализ нескольких баз данных.,На этот раз представим Базу данных ЭНКОРИ.,Одним из преимуществ этой базы данных является то, что она объединяет данные из нескольких баз данных.
База данных ЭНКОРИ
ENCORI (Энциклопедия интерактомов РНК) — это интегрированная база данных, в основном предназначенная для предоставления информации о взаимодействии между молекулами РНК, особенно данных о взаимодействии между микро РНК, дн РНК, м РНК и т. д.
Ключевые особенности и функции ENCORI:
- Сеть взаимодействия РНК: ENCORI предоставляет информацию о взаимодействии между различными молекулами РНК, включая дн РНК-ми РНК, микро РНК-м РНК, м РНК-м РНК и т. д. Исследователи могут использовать эти данные, чтобы лучше понять роль РНК в регуляции клеток. Объединив разнообразные экспериментальные данные и прогнозы биоинформатики, ENCORI может помочь выяснить, как молекулы РНК взаимодействуют, регулируя экспрессию генов.
- Анализ генов-мишеней микро РНК: база данных объединяет большое количество данных экспериментальной проверки и прогнозирования микро РНК и их генов-мишеней. Пользователи могут запрашивать целевые гены конкретной микро РНК или просматривать потенциальные регуляторные микро РНК гена. Функциональность особенно полезна при изучении регуляторной роли микро РНК при сложных заболеваниях, таких как рак.
- lncRNA Функция Примечание Пояснение: ENCORI предоставил lncRNA Интерпретация его функций, включая его потенциальные регуляторные механизмы и его роль в заболевании. Включено в базу данных lncRNA и miRNA и mRNA информация о взаимодействии, которая полезна для исследования lncRNA Функция и механизм имеют решающее значение.
- Интеграция данных мультиомики: ENCORI Интегрирует различные данные омики, включая данные транскриптома, эпигеномные данные, взаимодействия белков и т. д., для поддержки RNA Многоперспективный анализ взаимодействий. Пользователи могут проводить корреляционный анализ и исследования на основе этих данных. RNA Взаимодействие меняется в различных биологических условиях.
- Ассоциация болезней: ENCORI предоставил Различный RNA Информация об ассоциации молекул при заболеваниях, особенно раке RNA данные взаимодействия. Исследователи могут использовать этот анализ данных RNA Потенциальные механизмы действия молекул при сложных заболеваниях, таких как рак.
- Просмотр и визуализация данных: ENCORI Удобный интерфейс пользователя и богатые функции визуализации поддерживают быстрый просмотр, запрос и визуализацию данных. Пользователи могут легко найти то, что им интересно. RNA Информация о молекулах и взаимодействии и создание соответствующих диаграмм.

На этот раз мы пытаемся найти информацию о роли микро РНК и м РНК.
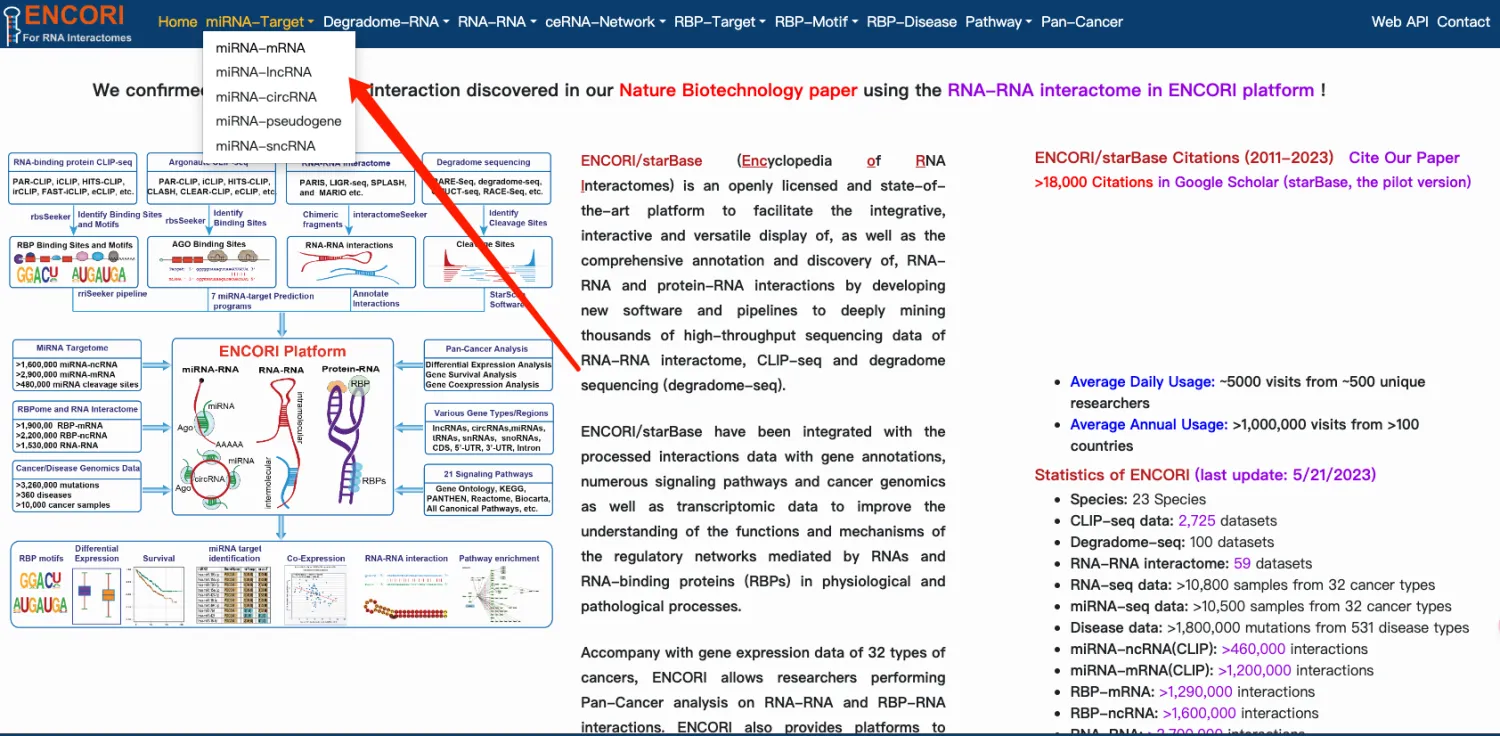
В левом интерфейсе вы можете ввести молекулы микро РНК, которые необходимо найти, а в правой части — скачать полные данные.

Разработчик также предоставляет веб-интерфейс, позволяющий загружать большие объемы данных одновременно. Если вам нужно загрузить все данные, вам необходимо изменить параметры. Эти параметры включают в себя:
- assembly=[genome версия]: укажите версию эталонного генома. Обычно используемые версии включают в себя hg38 (версия человеческого генома 38) и mm10 (версия генома мыши 10)。
- geneType=[main gene type]:
- Укажите основной тип гена для запроса. Варианты включают: м РНК, дн РНК, псевдоген, цирк РНК, мяк РНК.
- miRNA=[имя микро РНК]: укажите имя запрашиваемой микро РНК в таком формате, как hsa-miR-21-5p (hsa представляет собой микро РНК человека). Вы можете использовать «все» для запроса всех нормативных данных.
- clipExpNum=[integer]: указывает минимальное значение, которое поддерживает отношение управления CLIP-seq. CLIP-seq (Секвенирование иммунопреципитации с перекрестными связями) — экспериментальная технология взаимодействия РНК и белков. Этот параметр устанавливает нижний предел экспериментальных данных, который поддерживает регуляторные взаимосвязи.
- degraExpNum=[целое число]: указывает минимальное количество экспериментов degradome-seq, которые поддерживают нормативные отношения. Degradome-seq — это экспериментальный метод, используемый для обнаружения продуктов деградации РНК. Это значение установлено для обеспечения надежности экспериментального подтверждения данных.
- pancancerNum=[целое число]: укажите минимальное количество типов рака для проведения пан-ракового анализа (перекрестных типов рака). Необязательный диапазон — от 1 до 32, причем более высокие значения указывают на большее количество типов рака, поддерживаемых данными.
- programNum=[целое число]: указывает минимальное количество целевых программ прогнозирования. Выберите значение от 1 до 7 (максимум 7 программ), чтобы установить нормативные отношения, которые должны поддерживаться как минимум несколькими программами.
- program=[string]: укажите программу, используемую для прогнозирования целевых генов. Обычно используемые программы включают: PITA, RNA22, miRmap, DIANA-microT, miRanda, PicTar. и TargetScan。
- target=[имя гена]: укажите имя целевого гена, которое будет запрошено. Например, ТП53. Вы также можете использовать «все» для запроса всех нормативных данных.
- cellType=[тип клетки]: укажите тип клетки, например HeLa. Вы можете использовать «все», чтобы загрузить нормативные данные для всех типов клеток.

Шаг процесса
Откройте терминал, создайте скрипт и настройте путь.
# Создать скрипт
nano ENCORI.shВставьте содержимое
#!/bin/bash
# Определите желаемую процедуру прогнозирования
programs=("PITA" "RNA22" "miRmap" "DIANA-microT" "miRanda" "PicTar" "TargetScan")
# Загрузка данных для каждой программы в цикле
for prog in "${programs[@]}"
do
# Используйте Curl для загрузки данных и сохраните соответствующий файл в гостевом доме.
curl "https://rnasysu.com/encori/api/miRNATarget/?assembly=hg38&geneType=mRNA&miRNA=all&clipExpNum=5°raExpNum=0&pancancerNum=0&programNum=1&program=${prog}&target=all&cellType=all" > "ENCORI_hg38_CLIP-seq_${prog}.txt"
doneбегать
bash ./ENCOR.shФайл небольшой и загружается очень быстро.
Интегрируйте данные с R
1.Импорт
rm(list = ls())
# В ДИАНА-микро Т данных нет, поэтому при интеграции их необходимо удалить.
programs <- c("PITA","RNA22","miRmap",# "DIANA-microT",
"miRanda","PicTar","TargetScan")2. Предварительная обработка данных
data_list <- list()
for (i in programs) {
# Прочитать файл и сохранить R различные переменные в окружающей среде
data <- read.delim(paste0("ENCORI_hg38_CLIP-seq_", i, ".txt"),
sep = "\t", quote = "", row.names = NULL, comment.char = "#")
# использовать assign() Функция создает имя динамической переменной
assign(paste0("data_", i), data)
# Сохраните данные и имена столбцов в списке приезжать соответственно.
data_list[[i]] <- data
}
# проверить данные
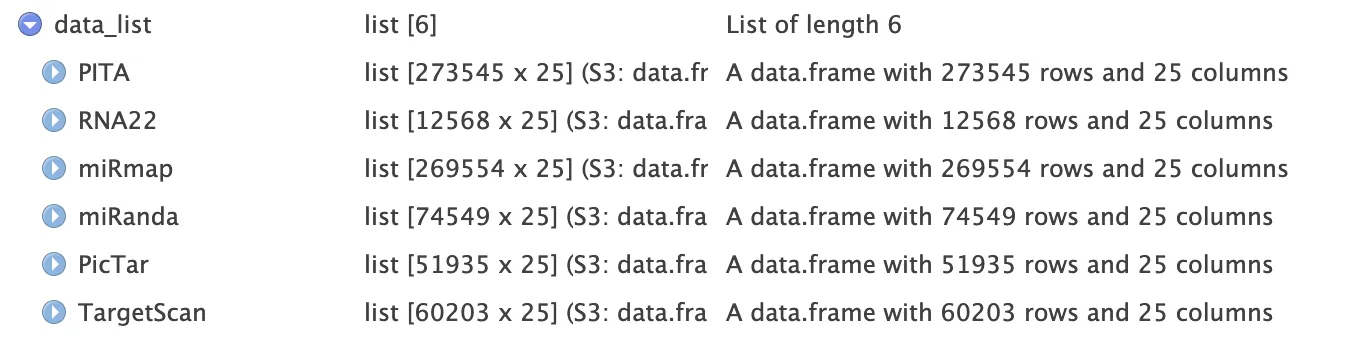
data_list[["PITA"]][1:4,1:4]
# miRNAid miRNAname geneID geneName
# 1 MIMAT0000062 hsa-let-7a-5p ENSG00000116237 ICMT
# 2 MIMAT0000062 hsa-let-7a-5p ENSG00000173641 HSPB7
# 3 MIMAT0000062 hsa-let-7a-5p ENSG00000244038 DDOST
# 4 MIMAT0000062 hsa-let-7a-5p ENSG00000007968 E2F2После этого вы можете просмотреть данные и построить диаграмму Венна.
Введите ключевое слово: 241019 в общедоступной учетной записи, чтобы получить загруженные данные ENCORI-miRNA (30 дней).
Ссылки:
- База данных ЭНКОРИ:https://rnasysu.com/encori/
- Дерево навыков биоксина: https://mp.weixin.qq.com/s/dkVBcls3f0acXtPnFSXeWw
- Группа новичков Шэнсиня: https://mp.weixin.qq.com/s/1voOEoFOQlym0GNl94dEEQ
Примечание:Если у вас есть сомнения по поводу содержания или вы обнаружили явные ошибки,,Пожалуйста, свяжитесь с серверной частью(Добро пожаловать для общения)。Доступно больше контентазакрывать Примечание Официальный аккаунт:Ковчег рождения
- END -

Неразрушающее увеличение изображений одним щелчком мыши, чтобы сделать их более четкими артефактами искусственного интеллекта, включая руководства по установке и использованию.

Копикодер: этот инструмент отлично работает с Cursor, Bolt и V0! Предоставьте более качественные подсказки для разработки интерфейса (создание навигационного веб-сайта с использованием искусственного интеллекта).

Новый бесплатный RooCline превосходит Cline v3.1? ! Быстрее, умнее и лучше вилка Cline! (Независимое программирование AI, порог 0)

Разработав более 10 проектов с помощью Cursor, я собрал 10 примеров и 60 подсказок.

Я потратил 72 часа на изучение курсорных агентов, и вот неоспоримые факты, которыми я должен поделиться!
Идеальная интеграция Cursor и DeepSeek API

DeepSeek V3 снижает затраты на обучение больших моделей

Артефакт, увеличивающий количество очков: на основе улучшения характеристик препятствия малым целям Yolov8 (SEAM, MultiSEAM).

DeepSeek V3 раскручивался уже три дня. Сегодня я попробовал самопровозглашенную модель «ChatGPT».

Open Devin — инженер-программист искусственного интеллекта с открытым исходным кодом, который меньше программирует и больше создает.

Эксклюзивное оригинальное улучшение YOLOv8: собственная разработка SPPF | SPPF сочетается с воспринимаемой большой сверткой ядра UniRepLK, а свертка с большим ядром + без расширения улучшает восприимчивое поле

Популярное и подробное объяснение DeepSeek-V3: от его появления до преимуществ и сравнения с GPT-4o.

9 основных словесных инструкций по доработке академических работ с помощью ChatGPT, эффективных и практичных, которые стоит собрать

Вызовите deepseek в vscode для реализации программирования с помощью искусственного интеллекта.

Познакомьтесь с принципами сверточных нейронных сетей (CNN) в одной статье (суперподробно)

50,3 тыс. звезд! Immich: автономное решение для резервного копирования фотографий и видео, которое экономит деньги и избавляет от беспокойства.

Cloud Native|Практика: установка Dashbaord для K8s, графика неплохая

Краткий обзор статьи — использование синтетических данных при обучении больших моделей и оптимизации производительности

MiniPerplx: новая поисковая система искусственного интеллекта с открытым исходным кодом, спонсируемая xAI и Vercel.

Конструкция сервиса Synology Drive сочетает проникновение в интрасеть и синхронизацию папок заметок Obsidian в облаке.

Центр конфигурации————Накос

Начинаем с нуля при разработке в облаке Copilot: начать разработку с минимальным использованием кода стало проще

[Серия Docker] Docker создает мультиплатформенные образы: практика архитектуры Arm64

Обновление новых возможностей coze | Я использовал coze для создания апплета помощника по исправлению домашних заданий по математике

Советы по развертыванию Nginx: практическое создание статических веб-сайтов на облачных серверах

Feiniu fnos использует Docker для развертывания личного блокнота Notepad

Сверточная нейронная сеть VGG реализует классификацию изображений Cifar10 — практический опыт Pytorch

Начало работы с EdgeonePages — новым недорогим решением для хостинга веб-сайтов

[Зона легкого облачного игрового сервера] Управление игровыми архивами


