Краткое изложение ресурсов общедоступных баз данных по протеомике
Что касается баз данных белков, то с ними все знакомы. По сути, каждая база данных будет оснащена веб-инструментом, позволяющим каждому запрашивать или выполнять простой анализ данных о белках. Ниже приведены некоторые известные базы данных протеомов:
- TrEMBL: Библиотека данных белков, поддерживаемая Европейским институтом биоинформатики (EMBL-EBI), предоставляет информацию автоматических аннотаций на основе программы UniProt.
- UniProt: Комплексная высококачественная библиотека белков, содержащая аннотированные вручную белки из UniProtKB/Swiss-Prot и аннотированные in silico белки из TrEMBL.
- Human Protein Atlas: Проект, направленный на анализ экспрессии и локализации всех белков человека в различных тканях.
- STRING: Библиотека данных и веб-ресурс, предоставляющий известные и прогнозируемые белок-белковые взаимодействия.
нобаза данных протеомане белокданные Библиотека,В основномProteomeXchangeальянс,Это открытая общедоступная платформа хранения данных.,Предназначен для хранилища и общих данных масс-спектрометрии (МС). Он состоит из нескольких библиотек протеомных данных.,Бесплатный архив PRIDE, MassIVE, PeptideAtlas, iProX и другие. д. 。 。
Например, мы можем увидеть статью по протеомике: https://www.sciencedirect.com/science/article/pii/S0300483X20302912?via%3Dihub.
Availability of data and material
The proteomics data have been deposited to the ProteomeXchange Consortium via the PRIDE (Perez-Riverol et al., 2019) partner repository with the dataset identifier PXD020248.
Целью ProteomeXchange является содействие стандартизации, совместному использованию и повторному использованию данных, а также поддержка дальнейшего развития исследований в области протеомики. Он следует принципам FAIR (находимость, доступность, совместимость и возможность повторного использования) для обеспечения качества и удобства использования данных. В целом мы знакомы с iProX (Международная платформа обмена протеомикой)иPRIDE (База данных протеомной идентификации)Вот и все:
- iProX: iProX (Международная платформа обмена протеомикой)Его установили китайские исследователи протеомики.данные Библиотека,Целью хранения является сохранение и обмен данными протеомики на основе масс-спектрометрии. iProX предоставляет платформу для исследователей,Для отправки, управления и доступа к протеомным данным,Поддержка стандартизации и обмена данными. Библиотека данных iProX также соответствует стандарту данных ProteomeXchange.,Облегчает интеграцию и анализ глобальной протеомики.
- PRIDE: PRIDE (База данных протеомной идентификации)это библиотека данных, поддерживаемая EMBL-EBI (Европейский институт биоинформатики).,Он является частью консорциума ProteomeXchange. ПРАЙД специализируется на сборе и хранении данных масс-спектрометрии.,Особенно идентификация и количественная информация о белках и пептидах. Библиотека данных PRIDE позволяет пользователям загружать данные и скачивать общедоступные коллекции данных.,и предоставляет инструмент для серии анализов,Помогите исследователям проводить протеомные исследования.
PRIDE (База данных протеомной идентификации)
это библиотека данных, поддерживаемая EMBL-EBI (Европейский институт биоинформатики).,Доступен по ссылке:https://www.ebi.ac.uk/pride/archive?sortDirection=DESC&page=2&pageSize=20
Просмотрите количество наборов данных, находящихся в настоящее время в базе данных: Список наборов данных (27273).
Например, недавний общедоступный набор данных таков: Миразомы нейтрофильного происхождения являются важной частью системы свертывания крови у мышей.
Вы можете ясно видеть, что база данных Pride предоставляет данные масс-спектрометрического прибора в необработанном формате и файлы матрицы экспрессии протеома для этого набора данных:
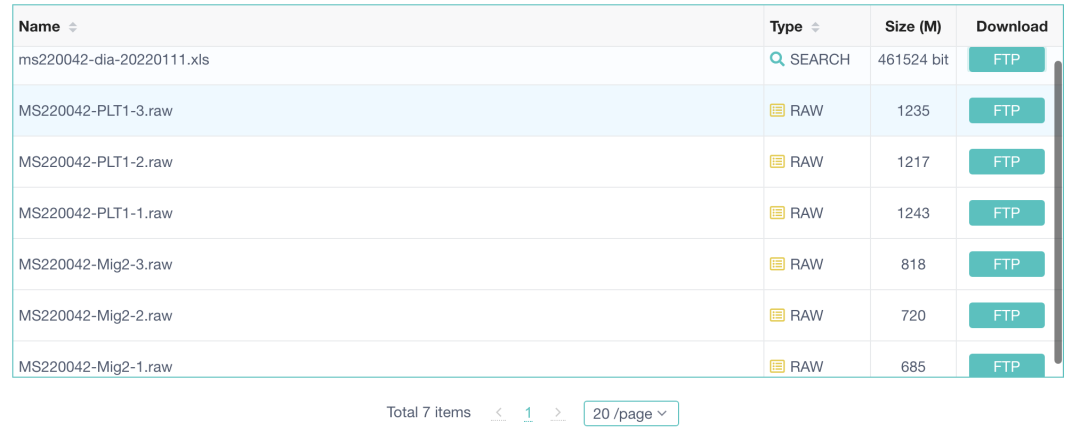
Набор данных дифференциального анализа мышей-две группы-протеома
Ссылка на подробный набор данных находится по адресу: https://www.ebi.ac.uk/pride/archive/projects/PXD051229.
Но обычно мы просто открываем внутреннюю частьms220042-dia-20220111.xlsфайл для последующего дифференциального анализа экспрессии протеома.Вот и все
iProX (Международная платформа обмена протеомикой)
существовать https://www.iprox.cn/page/BWV016.html Пока ты это видишь 3,676 entries,Потому что это библиотека данных, созданная китайскими исследователями протеомики.,Так что большая частьданные Все коллекции естьКитайские научные исследователипредоставил,И большинство из них - ГОРДОСТЬ (База данных протеомной идентификатор) также имеет идентификатор, как показано ниже;
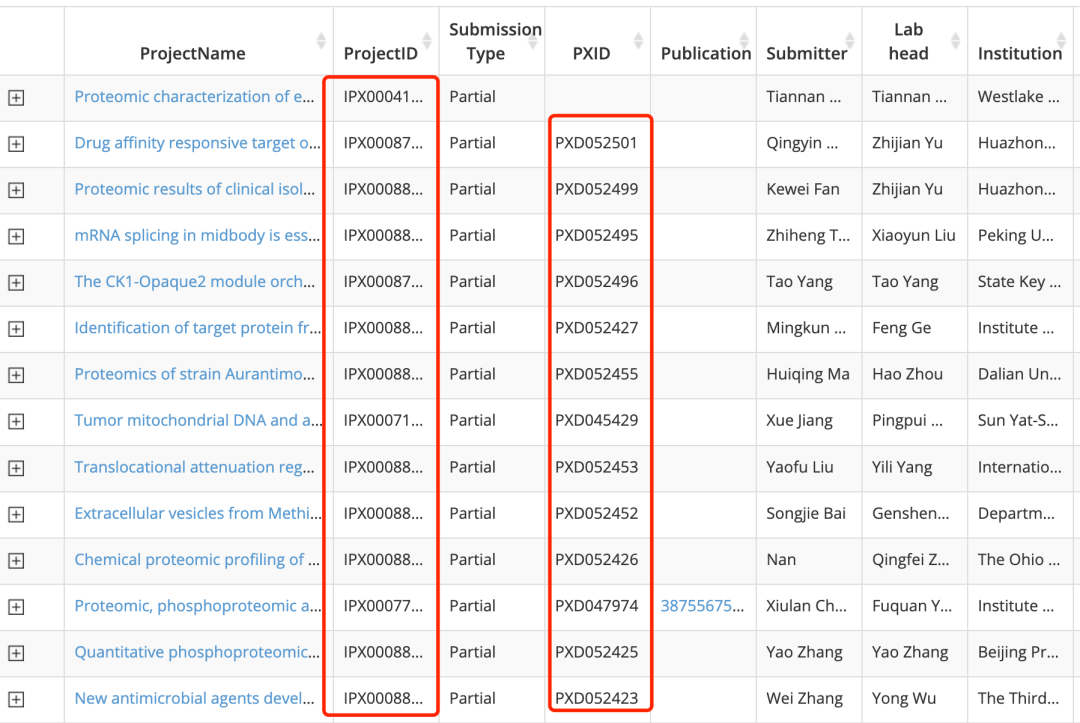
3,676 entries,
Нажмите на любой набор данных, чтобы войти: https://www.iprox.cn/page/ProjectFileList.html?projectId=IPX0006535000.
Proteome of Chinese Breast Cancers (FUSCC-Shao Lab)
IPX0006535000
Partial
PXD042886
Jiang YZ, Shao ZM. Molecular features and clinical implications of the heterogeneity in Chinese patients with HER2-low breast cancer. Nature Communications. 2023 Aug 22;14(1):5112-. doi:10.1038/s41467-023-40715-x.
Zhiming Shao
Zhiming Shao
Fudan University Shanghai Cancer Center
1
2023-06-11 08:40:12
Вы также можете увидеть другие его идентификаторы.,Вам нужен только идентификатор для доступа к записям данных в различных библиотеках данных.,например:
- https://proteomecentral.proteomexchange.org/cgi/GetDataset?ID=PXD042886
- https://www.ebi.ac.uk/pride/archive/projects/PXD042886
Если набор данных не содержит файла матрицы экспрессии белков
Поскольку существует множество различных технологических платформ протеомики, таких как Маркеры DIA, Label Free и TMT,их соответствующие Процесс предварительной обработки данных Есть некоторые различия:
Процесс предварительной обработки данных DIA (независимый от данных сбор):
- Стратегия поиска спектронавта:
- Используйте программное обеспечение Spectronaut для поиска в библиотеке и получения относительной количественной информации о пептидах и белках.
- данныеlog2Конвертировать、Фильтрация и заполнение отсутствующих значений、данныестандартизация,Возможно, используйте Combat, чтобы убрать пакетные эффекты.
- Стратегия поиска в базе данных ДИА-НН:
- Используйте DIA-NN для поиска в базе данных и получения относительных количественных значений.
- Выполнение преобразования log2, стандартизация данных и обработка пропущенных значений.,Наконец, были идентифицированы дифференциальные белки.
Процесс предварительной обработки данных без меток:
- Стратегия поиска в базе данных Maxquant:
- Результаты поиска в базе данных предоставляют три количественных значения: интенсивность, iBAQ и интенсивность LFQ.
- данныеlog2Конвертировать、Нормализация медианы или квантиля внутри выборки、Фильтрация и заполнение отсутствующих значений。
- Проведите количественный анализ различий.
- Стратегия поиска в базе данных Proteome Discoverer (PD):
- Значение количественного анализа по умолчанию — iBAQ.
- Метод стандартизации — FOT (доля общего количества).
- Чтобы заполнить недостающие значения, выберите соответствующий порог заполнения.
- Последующий анализ.
Процесс предварительной обработки данных TMT (Tandem Mass Tag):
- Стратегия поиска MSFragger:
- Используйте MSFragger для поиска в базе данных и получения файла результатов поиска в базе данных в формате pepXML.
- Количественное определение и фильтрацию пептидов, белков и посттрансляционных модификаций проводили с использованием пакета Philosopherинструмент.
- PeptideProphet выполняет идентификацию и проверку пептидов, а PTMProphet выполняет идентификацию сайта модификации.
- ProteinProphet используется для идентификации белков.
- Используйте Philosopher для фильтрации и количественного определения FDR, чтобы получить интенсивность репортерных ионов TMT.
- Коррекция выборки опорного канала для многократного преобразования и нормализации данных.
- преобразование log2, внутривыборочная медианная стандартизация, обработка пропущенных значений, удаление пакетного эффекта, анализ дифференциальных выражений.

Неразрушающее увеличение изображений одним щелчком мыши, чтобы сделать их более четкими артефактами искусственного интеллекта, включая руководства по установке и использованию.

Копикодер: этот инструмент отлично работает с Cursor, Bolt и V0! Предоставьте более качественные подсказки для разработки интерфейса (создание навигационного веб-сайта с использованием искусственного интеллекта).

Новый бесплатный RooCline превосходит Cline v3.1? ! Быстрее, умнее и лучше вилка Cline! (Независимое программирование AI, порог 0)

Разработав более 10 проектов с помощью Cursor, я собрал 10 примеров и 60 подсказок.

Я потратил 72 часа на изучение курсорных агентов, и вот неоспоримые факты, которыми я должен поделиться!
Идеальная интеграция Cursor и DeepSeek API

DeepSeek V3 снижает затраты на обучение больших моделей

Артефакт, увеличивающий количество очков: на основе улучшения характеристик препятствия малым целям Yolov8 (SEAM, MultiSEAM).

DeepSeek V3 раскручивался уже три дня. Сегодня я попробовал самопровозглашенную модель «ChatGPT».

Open Devin — инженер-программист искусственного интеллекта с открытым исходным кодом, который меньше программирует и больше создает.

Эксклюзивное оригинальное улучшение YOLOv8: собственная разработка SPPF | SPPF сочетается с воспринимаемой большой сверткой ядра UniRepLK, а свертка с большим ядром + без расширения улучшает восприимчивое поле

Популярное и подробное объяснение DeepSeek-V3: от его появления до преимуществ и сравнения с GPT-4o.

9 основных словесных инструкций по доработке академических работ с помощью ChatGPT, эффективных и практичных, которые стоит собрать

Вызовите deepseek в vscode для реализации программирования с помощью искусственного интеллекта.

Познакомьтесь с принципами сверточных нейронных сетей (CNN) в одной статье (суперподробно)

50,3 тыс. звезд! Immich: автономное решение для резервного копирования фотографий и видео, которое экономит деньги и избавляет от беспокойства.

Cloud Native|Практика: установка Dashbaord для K8s, графика неплохая

Краткий обзор статьи — использование синтетических данных при обучении больших моделей и оптимизации производительности

MiniPerplx: новая поисковая система искусственного интеллекта с открытым исходным кодом, спонсируемая xAI и Vercel.

Конструкция сервиса Synology Drive сочетает проникновение в интрасеть и синхронизацию папок заметок Obsidian в облаке.

Центр конфигурации————Накос

Начинаем с нуля при разработке в облаке Copilot: начать разработку с минимальным использованием кода стало проще

[Серия Docker] Docker создает мультиплатформенные образы: практика архитектуры Arm64

Обновление новых возможностей coze | Я использовал coze для создания апплета помощника по исправлению домашних заданий по математике

Советы по развертыванию Nginx: практическое создание статических веб-сайтов на облачных серверах

Feiniu fnos использует Docker для развертывания личного блокнота Notepad

Сверточная нейронная сеть VGG реализует классификацию изображений Cifar10 — практический опыт Pytorch

Начало работы с EdgeonePages — новым недорогим решением для хостинга веб-сайтов

[Зона легкого облачного игрового сервера] Управление игровыми архивами


