Код всего процесса менделевского промежуточного анализа (1)

фон:Это наблюдается,Более высокий уровень обучения связан с меньшим риском болезни Альцгеймера. Однако,Биологические механизмы, лежащие в основе этой ассоциации, остаются неясными. Защитный эффект обучения против болезни Альцгеймера может быть опосредован увеличением резерва мозга.
метод:Для исследования мы используем двухвыборочную менделевскую рандомизацию.обучатьстепень、Предполагаемая причинно-следственная связь между структурным резервом мозга, представленным фенотипами МРТ, и болезнью Альцгеймера。Мы сделали заказпеременная Менделевский рандомизационный анализ,Изучить двунаправленную связь между (i) степенью обучения и болезнью Альцгеймера; (ii) степенью обучения и фенотипом визуализации и (iii) фенотипом визуализации и болезнью Альцгеймера; Множественная переменная менделевская рандомизация использовалась для оценки того, опосредуют ли структурные фенотипы мозга влияние обучения на риск болезни Альцгеймера.
результат:нашрезультатподдерживатьобучатьстепень对阿尔茨海默病风险的保护性因果效应,и потенциальные двунаправленные причинно-следственные связи между степенью обученности и макро- и микроструктурой мозга. Однако,Мы не нашли доказательств того, что эти структурные маркеры влияют на риск болезни Альцгеймера. обучать Степень защиты от болезни Альцгеймера может быть достигнута за счет других мер резерва мозга, не включенных в это исследование.,или опосредовано другими механизмами.
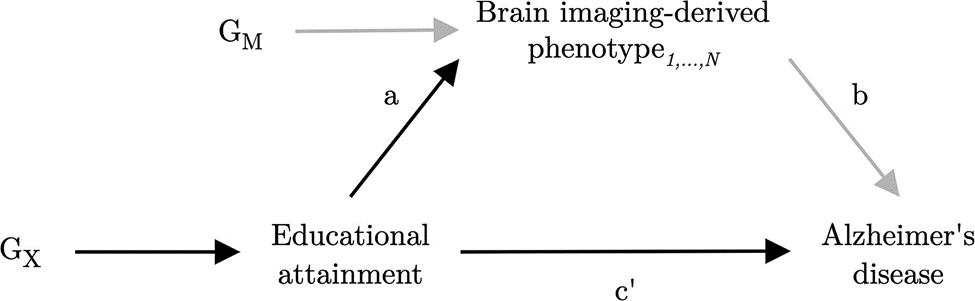
GX представляет собой набор инструментальных переменных советника. Параметр a представляет собой прямое причинное влияние ЭА на фенотипы 1, ..., N, полученные при визуализации, а параметр b представляет прямое причинное влияние фенотипов 1, ..., N, полученных при визуализации, на болезнь Альцгеймера. Параметр c' представляет собой прямое причинное влияние ЭА на болезнь Альцгеймера. Параметры a и c' оцениваются с использованием набора переменных GX, а параметр b оценивается с использованием набора переменных GM. Смешивающие переменные не показаны на рисунке.
Примерно уяснив идею статьи, давайте посмотрим, есть ли что-то, что нас больше всего беспокоит——
R code for reproducing all MR analyses can be found on https://github.com/as2970/EA_brain_AD_MR.
чудесный! ! ! Спасибо автору статьи~
основной код шага 0
library(tidyverse)
library(data.table)
# library(ivpack)
library(meta)
library(devtools)
library(pacman)
library(TwoSampleMR)
library(MRInstruments)
library(ieugwasr)
# install_github("phenoscanner/phenoscanner")
library(phenoscanner)
library(LDlinkR)
library(mr.raps)
library(MRPRESSO)
library(extrafont)
library(anchors)
Здесь автор написал функцию, если вы когда-нибудь
Введение в биокредит&данные Откройте для себя онлайн-классы в прямом эфире6月班
Блок интеллектуального анализа данных GEO, прошедший обучение на марафоне
Если у вас есть определенные знания языка R, вы можете попытаться понять эту функцию. Последующий анализ будет значительно упрощен⬇.
## MR_prep <- function(exp, out) {
# Extracting instruments
exp_dat <- read_exposure_data(
filename = paste0(deparse(substitute(exp)), "_exposure.txt", sep = ""),
clump = FALSE,
sep = " ",
snp_col = "SNP",
beta_col = "BETA",
se_col = "SE",
eaf_col = "AF1",
effect_allele_col = "A1",
other_allele_col = "A2",
pval_col = "P",
ncase_col = "N_case",
ncontrol_col = "N_control",
samplesize_col = "N",
min_pval = 1e-200,
log_pval = FALSE,
chr_col = "CHR",
pos_col = "POS"
)
# Clumping instruments
exp_dat <- exp_dat %>%
rename(
rsid = SNP,
pval = pval.exposure
)
exp_dat_clumped <- ld_clump(
dat = exp_dat,
clump_kb = 10000,
clump_r2 = 0.001,
clump_p = 5e-8,
plink_bin = genetics.binaRies::get_plink_binary(),
bfile = "" #path to LD reference dataset
)
exp_dat_clumped <- exp_dat_clumped %>%
rename(
SNP = rsid,
pval.exposure = pval
)
# Printing number of IVs for exposure
print(paste0("Number of IVs: ", as.character(length(exp_dat_clumped$SNP))))
Чтение конечных данных
# Extracting instruments from outcome GWAS
out_dat <- read_outcome_data(
filename = paste0(deparse(substitute(out)), "_outcome.txt", sep = ""),
snps = exp_dat_clumped$SNP,
sep = " ",
snp_col = "SNP",
beta_col = "BETA",
se_col = "SE",
eaf_col = "AF1",
effect_allele_col = "A1",
other_allele_col = "A2",
pval_col = "P",
ncase_col = "N_case",
ncontrol_col = "N_control",
samplesize_col = "N",
min_pval = 1e-200,
log_pval = FALSE,
chr_col = "CHR",
pos_col = "POS"
)
# Identifying & printing exposure instruments missing from outcome GWAS
missing_IVs <- exp_dat_clumped$SNP[!(exp_dat_clumped$SNP %in% out_dat$SNP)]
print(paste0("Number of IVs missing from outcome GWAS: ", as.character(length(missing_IVs))))
print("List of IVs missing from outcome GWAS:")
for (i in 1:length(missing_IVs)) {
print(paste0(missing_IVs[i]))
}
Поиск прокси-SNP
# Replacing missing instruments from outcome GWAS with proxies
if(length(missing_IVs) == 0) {
print("All exposure IVs found in outcome GWAS.")
} else {
print("Some exposure IVs missing from outcome GWAS.")
out_full <- fread(paste0(deparse(substitute(out)), "_outcome.txt", sep = ""))
for (i in 1:length(missing_IVs)) {
proxies <- LDproxy(snp = missing_IVs[i], pop = "EUR", r2d = "r2", token = "6fb632e022ef", file = FALSE)
proxies <- proxies[proxies$R2 > 0.8, ]
proxy_present = FALSE
if(length(proxies$RS_Number) == 0){
print(paste0("No proxy SNP available for ", missing_IVs[i]))
} else {
for (j in 1:length(proxies$RS_Number)) {
proxy_present <- proxies$RS_Number[j] %in% out_full$SNP
if (proxy_present) {
proxy_SNP = proxies$RS_Number[j]
proxy_SNP_allele_1 = str_sub(proxies$Alleles[j], 2, 2)
proxy_SNP_allele_2 = str_sub(proxies$Alleles[j], 4, 4)
original_SNP_allele_1 = str_sub(proxies$Alleles[1], 2, 2)
original_SNP_allele_2 = str_sub(proxies$Alleles[1], 4, 4)
break
}
}
}
if(proxy_present == TRUE) {
print(paste0("Proxy SNP found. ", missing_IVs[i], " replaced with ", proxy_SNP))
proxy_row <- out_dat[1, ]
proxy_row$SNP = missing_IVs[i]
proxy_row$beta.outcome = as.numeric(out_full[out_full$SNP == proxy_SNP, "BETA"])
proxy_row$se.outcome = as.numeric(out_full[out_full$SNP == proxy_SNP, "SE"])
if (out_full[out_full$SNP == proxy_SNP, "A1"] == proxy_SNP_allele_1) proxy_row$effect_allele.outcome = original_SNP_allele_1
if (out_full[out_full$SNP == proxy_SNP, "A1"] == proxy_SNP_allele_2) proxy_row$effect_allele.outcome = original_SNP_allele_2
if (out_full[out_full$SNP == proxy_SNP, "A2"] == proxy_SNP_allele_1) proxy_row$other_allele.outcome = original_SNP_allele_1
if (out_full[out_full$SNP == proxy_SNP, "A2"] == proxy_SNP_allele_2) proxy_row$other_allele.outcome = original_SNP_allele_2
proxy_row$pval.outcome = as.numeric(out_full[out_full$SNP == proxy_SNP, "P"])
proxy_row$samplesize.outcome = as.numeric(out_full[out_full$SNP == proxy_SNP, "N"])
if("N_case" %in% colnames(out_full)) proxy_row$ncase.outcome = as.numeric(out_full[out_full$SNP == proxy_SNP, "N_case"])
if("N_control" %in% colnames(out_full))proxy_row$ncontrol.outcome = as.numeric(out_full[out_full$SNP == proxy_SNP, "N_control"])
proxy_row$chr.outcome = as.numeric(exp_dat_clumped[exp_dat_clumped$SNP == missing_IVs[i], "chr.exposure"])
proxy_row$pos.outcome = as.numeric(exp_dat_clumped[exp_dat_clumped$SNP == missing_IVs[i], "pos.exposure"])
if("AF1" %in% colnames(out_full)) proxy_row$eaf.outcome = as.numeric(out_full[out_full$SNP == proxy_SNP, "AF1"])
out_dat <- rbind(out_dat, proxy_row)
}
if(proxy_present == FALSE) {
print(paste0("No proxy SNP available for ", missing_IVs[i], " in outcome GWAS."))
}
}
}
затем гармонизировать
# Harmonising exposure and outcome datasets
dat <- harmonise_data(
exposure_dat = exp_dat_clumped,
outcome_dat = out_dat,
action = 2
)
## } ##Конец функции самостоятельного написания
Эта функция очень длинная,Поначалу может показаться немного трудным смотреть,Не волнуйтесь,Используйте проблемы с пользой??функция`,Все желающие также могут обсудить и решить проблему в комментариях~
напримерread_exposure_dataфункция,Когда мы готовимся предоставить данные, мы должны учитывать тип данных, требуемых этой функцией.,Вот и все??read_exposure_dataВзгляните,В основном смотримArguments,Что необходимо,Какие данные могут оказаться ненужными.

??read_outcome_dataфункция То же самое верно,Это то, что нам нужно знать заранее, на этапе подготовки! ! ! Все нужно делать заранее (Урок редактора...)
Советы
Что касается конвертации eaf и maf, то это небольшая деталь, но не стоит быть небрежным~
Поместите это здесь для справки
【https://www.bilibili.com/video/BV1De4y1577E/?vd_source=e5e36ce10569a7a5d647d18bdb42e4b5】
for (i in 1:length(BRCA1_TN$eaf_col)) {
if (BRCA1_TN$eaf_col[i]<0.5) {BRCA1_TN$eaf_col[i]= 1 -BRCA1_TN$eaf_col[i]
}else{BRCA1_TN$eaf_col[i] = BRCA1_TN$eaf_col[i]}
}
Нужно выполнить 4 шага кода, давайте идти шаг за шагом~

Неразрушающее увеличение изображений одним щелчком мыши, чтобы сделать их более четкими артефактами искусственного интеллекта, включая руководства по установке и использованию.

Копикодер: этот инструмент отлично работает с Cursor, Bolt и V0! Предоставьте более качественные подсказки для разработки интерфейса (создание навигационного веб-сайта с использованием искусственного интеллекта).

Новый бесплатный RooCline превосходит Cline v3.1? ! Быстрее, умнее и лучше вилка Cline! (Независимое программирование AI, порог 0)

Разработав более 10 проектов с помощью Cursor, я собрал 10 примеров и 60 подсказок.

Я потратил 72 часа на изучение курсорных агентов, и вот неоспоримые факты, которыми я должен поделиться!
Идеальная интеграция Cursor и DeepSeek API

DeepSeek V3 снижает затраты на обучение больших моделей

Артефакт, увеличивающий количество очков: на основе улучшения характеристик препятствия малым целям Yolov8 (SEAM, MultiSEAM).

DeepSeek V3 раскручивался уже три дня. Сегодня я попробовал самопровозглашенную модель «ChatGPT».

Open Devin — инженер-программист искусственного интеллекта с открытым исходным кодом, который меньше программирует и больше создает.

Эксклюзивное оригинальное улучшение YOLOv8: собственная разработка SPPF | SPPF сочетается с воспринимаемой большой сверткой ядра UniRepLK, а свертка с большим ядром + без расширения улучшает восприимчивое поле

Популярное и подробное объяснение DeepSeek-V3: от его появления до преимуществ и сравнения с GPT-4o.

9 основных словесных инструкций по доработке академических работ с помощью ChatGPT, эффективных и практичных, которые стоит собрать

Вызовите deepseek в vscode для реализации программирования с помощью искусственного интеллекта.

Познакомьтесь с принципами сверточных нейронных сетей (CNN) в одной статье (суперподробно)

50,3 тыс. звезд! Immich: автономное решение для резервного копирования фотографий и видео, которое экономит деньги и избавляет от беспокойства.

Cloud Native|Практика: установка Dashbaord для K8s, графика неплохая

Краткий обзор статьи — использование синтетических данных при обучении больших моделей и оптимизации производительности

MiniPerplx: новая поисковая система искусственного интеллекта с открытым исходным кодом, спонсируемая xAI и Vercel.

Конструкция сервиса Synology Drive сочетает проникновение в интрасеть и синхронизацию папок заметок Obsidian в облаке.

Центр конфигурации————Накос

Начинаем с нуля при разработке в облаке Copilot: начать разработку с минимальным использованием кода стало проще

[Серия Docker] Docker создает мультиплатформенные образы: практика архитектуры Arm64

Обновление новых возможностей coze | Я использовал coze для создания апплета помощника по исправлению домашних заданий по математике

Советы по развертыванию Nginx: практическое создание статических веб-сайтов на облачных серверах

Feiniu fnos использует Docker для развертывания личного блокнота Notepad

Сверточная нейронная сеть VGG реализует классификацию изображений Cifar10 — практический опыт Pytorch

Начало работы с EdgeonePages — новым недорогим решением для хостинга веб-сайтов

[Зона легкого облачного игрового сервера] Управление игровыми архивами


